Tras la publicación hace solo tres meses del primero de esta serie (relativo a dupilumab, comentado en esta sección de Panorama del número 451), se acaba de divulgar un segundo informe de evaluación de los medicamentos incluidos en el novedoso Sistema de Información para determinar el Valor Terapéutico en la Práctica Clínica Real de los Medicamentos de Alto Impacto Sanitario y Económico en el Sistema Nacional de Salud (VALTERMED, creado en 2018), para los que se ha establecido un protocolo clínicos de uso.
Se trata del referente a inotuzumab ozogamicina, un fármaco conjugado compuesto por un anticuerpo monoclonal anti-CD22 humano, inotuzumab, que está enlazado covalentemente con la molécula pequeña N-acetil-γ-calicheamicina, un producto natural semisintético citotóxico. Así, se dirige específicamente contra las células que expresan CD22 en su superficie, incluyendo las células cancerosas pro-B y pre-B, y tras su internalización, libera el derivado de calicheamicina, cuya activación induce roturas del ADN bicatenario de la célula cancerígena, la interrupción del ciclo y la muerte celular por apoptosis. El medicamento, designado como huérfano, fue comercializado en 2019 en nuestro país, con autorización para el tratamiento por vía intravenosa de adultos con leucemia linfoblástica aguda (LLA) de precursores de linfocitos B positivos para CD22 recidivante o refractaria; aquellos pacientes positivos para el cromosoma Filadelfia deben tener fracaso terapéutico con al menos un inhibidor de la tirosina-cinasa.
Desde enero de 2020 y hasta el 31 de diciembre de 2021, se habían registrado datos de 67 pacientes con LLA, la mayoría de ellos residentes en Madrid (16; 24%), Andalucía (13; 19%), País Vasco y Cataluña (8; 12% en los dos casos). Iniciaron tratamiento con inotuzumab ozogamicina 49 de ellos (73%), destacando entre sus características basales una media de edad de 38 años (5 eran menores de 18 años) y que el 54% eran mujeres. El protocolo farmacoclínico recoge que se realizarán al menos dos evaluaciones de la respuesta –estudio de médula ósea morfológico y por citometría de flujo para CD22– hasta progresión de la enfermedad o según la práctica clínica local, una inicial (tras 1 mes), otra a los 2 meses y a los 3 meses, para posteriormente espaciarlas cada 3 meses durante 1 año y cada 6 meses durante el segundo año.
Los resultados relativos a los 41 pacientes a los que se realizó la primera evaluación reflejan una tasa de respuesta del 71%, objetivada en 29 pacientes que responden favorablemente como respuesta completa (29,3%) o respuesta completa con enfermedad mínima residual (29,3%); en cambio, 11 pacientes no responden al tratamiento (42%), y 1 paciente fallece en este intervalo temporal. La segunda evaluación, realizada en 24 pacientes, apunta a una tasa de respuesta del 63%, registrándose otras 2 nuevas muertes y 2 recaídas. En evaluaciones posteriores se reduce progresivamente la disponibilidad de datos clínicos por recaídas o fallecimientos (11 pacientes en la tercera y solo 2 en la cuarta); hasta 16 pacientes solo han sido evaluados una vez. Considerando los datos en conjunto, y según la última evaluación de cada paciente (se dispone de datos para 42), se ha reportado una tasa de respuesta del 57% (24/42), mientras que no responden un 26% (11/42), están en recaída respecto a la anterior visita un 7% (3/42) y fallecen un 10% (4/42). Se registraron un total de 29 fallecidos.
Cabe destacar que el objetivo último del uso de inotuzumab ozogamicina es posibilitar la realización posterior de un trasplante alogénico de progenitores hematopoyéticos (alo-TPH) en los pacientes que obtienen una remisión completa, lo que se consiguió en 16 de los 49 pacientes que inician tratamiento (33%). Se ha estimado que la mediana de supervivencia de los 44 pacientes para los que se tienen datos es de 33,7 semanas (7,8 meses), cifra muy similar a la obtenida en el ensayo clínico que sustentó su autorización, donde alcanzó una mediana de 7,7 meses (vs. 6,5 meses con los regímenes de quimioterapia seleccionados como control). Si solo se considera a los pacientes que han recibido un alo-TPH y siguen vivos a la fecha de corte, la media de supervivencia es de más del doble: 75,8 semanas. La proporción de pacientes supervivientes a los 2 años alcanzó el 26% (IC95% 11,5-38,1), cifra ligeramente superior a la reportada en el ensayo clínico (22,6%).
En cuanto a la seguridad, se han notificado efectos adversos graves relacionados con el tratamiento en un total de 31 pacientes (63%), entre ellos 2 menores de 18 años, si bien solo se han registrado datos de efectos adversos en 40 de los pacientes (82% de los 49 que inician tratamiento). Los efectos adversos notificados con mayor frecuencia han sido trombocitopenia (65%) y neutropenia (55%). Así pues, en consonancia con la ficha técnica, los efectos adversos relacionados con trastornos de la sangre y del sistema linfático han sido los más comunes.
Pese a las limitaciones en la evidencia (relativas, por ejemplo, al número o características demográficas de los pacientes) que limitan la comparación directa entre el ensayo clínico y la práctica en vida real reflejada en VALTERMED, los resultados de efectividad parecen interesantes, en la línea de lo que se podía esperar por los datos pre-autorización. No obstante, no se pueden extraer conclusiones sólidas hasta que se tengan datos de más pacientes y a más largo plazo.
Archive
Revista PAM: 454
Número 454, Junio 2022
Aproximación al consumo de recursos sanitarios y al coste económico del manejo de pacientes con infección por SARS-CoV-2: un estudio retrospectivo
A pesar de contar con un Sistema Nacional de Salud (SNS) caracterizado por su cobertura universal, España es uno de los países que ha sufrido un mayor impacto a causa de la pandemia del SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus 2), según indican los datos de mortalidad y el descenso del Producto Interior Bruto (PIB), en comparación con otros países europeos. Por tanto, para evaluar con precisión los aspectos económicos relacionados con el SNS y la pandemia, se hace necesario generar evidencia sobre los recursos sanitarios empleados en el manejo de pacientes con COVID-19 y los costes asociados a estos. Sin embargo, este tipo de análisis en España resultan complejos debido a la escasez de evaluaciones económicas publicadas al respecto, así como a la variabilidad de los escasos costes estimados. Ante esta realidad, Calderón-Moreno y colaboradores plantearon un análisis retrospectivo de 9.811 casos con COVID-19, desde el principio de la pandemia hasta el 30 de junio de 2020, para estimar su impacto sobre el SNS.
El estudio dividió a los pacientes en dos cohortes: la primera, compuesta por 6.286 pacientes manejados íntegramente en atención primaria en Aragón y la segunda, con 3.525 casos de COVID-19, se tomó de un registro de pacientes del Hospital Universitario La Paz de Madrid, con información de los pacientes que requirieron ingreso hospitalario. De todos los casos incluidos en el estudio se registraron sus características demográficas (edad, género, origen y comorbilidades de riesgo) para la realización de subanálisis.
Para la estimación de costes se tomaron los siguientes datos: 1) manejo del paciente, diferenciando entre atención primaria, atención hospitalaria sin ingreso en la Unidad de Cuidados Intensivos (UCI), e ingreso hospitalario en UCI; 2) número de escáneres de rayos X y de tomografía axial computarizada (TAC) realizados; y 3) frecuencia de complicaciones de la infección por SARS-CoV-2 (infección respiratoria, arritmias, pancreatitis, enfermedades psiquiátricas, etc.). Todos estos parámetros fueron cuantificados mediante los costes unitarios publicados por el Ministerio de Sanidad y otras entidades públicas.
Los autores estimaron que el 59,13% de los pacientes con COVID-19 fueron derivados a atención primaria, frente al 40,87% que acudió a urgencias en el hospital y necesitó algún tipo de manejo hospitalario. De estos últimos, el 95,50% requirió ingreso fuera de la UCI, a diferencia del 4,50% restante, que, además, fue ingresado en la UCI. El coste promedio por paciente manejado en atención primaria fue de 729,79€. En el caso del paciente con ingreso hospitalario en planta, el coste estimado fue de 4.294,36€, cifra que ascendió a 14.440,68€ cuando el paciente precisó ingreso en UCI. Calculado como promedio ponderado, el coste del manejo de cada paciente que sufrió infección por SARS-CoV-2 fue de 2.373,24€. De este último coste, los escáneres de rayos X y TAC supusieron 27,17€ y 11,90€, respectivamente. Por su parte, el manejo de las complicaciones representó el 72% del coste total (1.707,66€), siendo el fallo renal la complicación más frecuente de la COVID-19, presente en el 8,46% de los casos, y con mayor impacto económico.
La evaluación económica se acompañó de distintos análisis en función de las características demográficas de los pacientes y comorbilidades. Todas ellas se asociaron con un incremento en el número de escáneres de rayos X necesarios y, por ende, sobre el coste total. El asma fue la única comorbilidad cuya evidencia respecto al incremento en el consumo de escáneres de rayos X no fue estadísticamente significativa.
En conclusión, el coste de los pacientes que sufren infección por SARS-CoV-2 se incrementa notablemente cuando el paciente acude al hospital y, aún más, cuando es ingresado en UCI. Además, las complicaciones aumentan notablemente el coste del manejo de estos pacientes, al igual que los escáneres de rayos X y TAC. En el caso concreto de estos últimos, algunas comorbilidades crónicas (diabetes, hipertensión, tabaquismo, asma, enfermedades cardíacas crónicas y/o bronquitis crónicas) se asocian a un consumo superior de recursos, incrementando a su vez el coste del manejo de la COVID-19.
Comentario
Este análisis estimó el coste promedio por paciente con COVID-19 en España en 2.373,24€. Además, pone de relieve el incremento pronunciado del coste en función de los requerimientos asistenciales del paciente, de forma que el asociado al abordaje desde atención primaria se multiplica por 20 cuando el paciente ingresa en la UCI.
Sin embargo, no se debe obviar que existen diversos factores que podrían variar esta estimación de costes. Por un lado, debido al conocimiento sobre el virus y su abordaje terapéutico generado tras las distintas oleadas de la pandemia, cabría esperar que el coste del manejo de la infección se haya visto reducido. Sin embargo, la gran variabilidad de los costes publicados por la Administración Sanitaria en función de la gravedad de las complicaciones, no haber considerado los tratamientos farmacológicos empleados frente a la infección y sus síntomas, o no haber monetizado las complicaciones a largo plazo (o aquellas acontecidas durante la estancia hospitalarias) son algunos factores que podrían aumentar el coste del manejo de la enfermedad, al igual que no haber contemplado los costes indirectos ocasionados por la pérdida de productividad de los pacientes.
En definitiva, queda patente que, si bien este estudio es un gran avance en la caracterización del impacto económico que la pandemia ha producido en el Sistema Sanitario español, sigue siendo preciso disponer de más estudios que generen una visión más amplia de la repercusión económica de la pandemia, a corto, medio y largo plazo.
Análisis coste-beneficio de la estrategia de cribado masivo de pacientes con COVID-19 asintomáticos en el área metropolitana norte de Barcelona
Una de las medidas tomadas para afrontar la pandemia por COVID-19 fue la detección temprana de casos de infección por SARS-CoV-2. Para ello, entre las estrategias disponibles, el uso del cribado masivo de individuos asintomáticos fue muy extendido a nivel global. En el caso de Cataluña, el cribado masivo se llevó a cabo mediante la realización de test de antígenos o PCR (Polimerase Chain Reaction), lo que permitió detectar casos precozmente, aislar a estos pacientes y cortar la cadena de contagio. No obstante, estas estrategias fueron ampliamente criticadas por las limitaciones en la evidencia sobre su efectividad, así como por su falta de planificación y el enorme coste que suponían para el Sistema Nacional de Salud (SNS).
En este contexto, López Seguí y colaboradores trataron de estimar la eficiencia del cribado masivo en Barcelona, durante la primera y segunda ola de la pandemia, por medio de un análisis coste-beneficio (ACB). Estos análisis son evaluaciones económicas que analizan los costes en relación con los resultados de una intervención sanitaria, con la particularidad de que los resultados se miden en unidades monetarias.
El ACB se realizó con los datos de 78 cribados masivos realizados desde el inicio de la pandemia hasta finalizar 2020, que totalizaron 125.865 individuos, de los que 1.719 fueron positivos a la infección por SARS-CoV-2 (1,37%). Para estimar los costes derivados de la intervención se consideró el número de test realizados, el número de rastreadores de contactos, la frecuencia de hospitalización e ingreso en UCI por COVID-19 y la frecuencia de aparición de secuelas de la infección a largo plazo. Posteriormente, se cuantificaron estos parámetros en base a sus costes unitarios y, además, se consideró el coste del seguimiento del paciente en cuarentena mediante llamadas telefónicas. Por su parte, la eficacia del cribado masivo se determinó en base a la tasa de positividad, número de contactos por individuo, proporción de contactos cercanos con individuos positivos a SARS-CoV-2, proporción de pacientes detectados antes de haber contagiado, número reproductivo (Ro), letalidad y frecuencia de aparición de secuelas de la infección. Finalmente, se calculó la pérdida de años de vida ajustados por calidad (AVACs) asociados a la secuela por COVID-19 y a la mortalidad, que se monetizó considerando que un AVAC equivale a 25.000€.
Los autores estimaron un coste de 8.372.265€ asociado a la estrategia de cribado masivo, de los cuales el 88% tuvo origen en los propios test empleados para el diagnóstico. La intervención detectó 1.719 casos de COVID-19, lo que se tradujo en la prevención de 5.429 infecciones, 168 hospitalizaciones, 111 ingresos en UCI, 56 casos de secuelas a largo plazo y 33 muertes. Estos resultados supusieron un ahorro de 3.762.322€, cifra que asciende a 10.028.690€ si se tiene en cuenta el valor monetario de los 156 y 95 AVACs asociados a la morbilidad a largo plazo y mortalidad, respectivamente, cuya pérdida se evitó gracias a la intervención.
Los resultados expuestos anteriormente permitieron estimar el ratio coste-beneficio (RCB) del cribado masivo de la infección por SARS-CoV-2. Este ratio refleja el retorno social generado por dicha intervención cuando se invierte 1€ en la misma, entendiendo el concepto de retorno social como el beneficio, expresado en términos económicos, que no se aprecia de forma directa en las ganancias obtenidas de una inversión. De esta forma, sin incluir el valor monetario de los AVACs, el RCB fue de 0,45, equivalente a un retorno social de 45 céntimos por cada euro invertido, lo que indicaría que la realización de cribados masivos de asintomáticos con COVID-19 no sería una intervención eficiente. En contrapartida, el RCB o retorno social resultante por cada euro invertido fue de 1,20, cuando se tomó en consideración el valor monetario de los AVACs. En este último análisis, se podría concluir que sería aconsejable realizar cribados masivos de la población para identificar precozmente casos de infección por SARS-CoV-2.
En resumen, los cribados masivos de pacientes asintomáticos con SARS-CoV-2 podrían generar importantes retornos sociales cuando se considera el impacto de la intervención sobre la calidad de vida relacionada con la salud (CVRS), considerando los AVACs. Se trata de una estrategia cuyo beneficio se ve maximizado cuando se emplea en poblaciones concretas, entre las que cabe destacar los pacientes de alto riesgo de desarrollar COVID-19 o aquellos en los que se espere una alta tasa de positividad.
Comentario
En primer lugar, resulta interesante apreciar cómo la realización del cribado masivo en todos los individuos asintomáticos implica un coste elevado, con el consiguiente coste de oportunidad asociado, pudiendo no ser una estrategia eficiente en ciertos casos. Los autores del citado estudio exponen la necesidad de dirigir cada intervención a la población que más se beneficiaría de ella, en este caso a pacientes donde se espere una tasa de positividad elevada, en pro de garantizar la sostenibilidad del sistema.
Más allá de este resultado, el estudio también pone en relieve la importancia de considerar la repercusión de una intervención sobre la calidad de vida relacionada con la salud de los pacientes, aspecto clave para determinar si dicha intervención es o no eficiente.
En esta misma línea, además del retorno social, el cribado masivo de asintomáticos potencialmente infectados con SARS-CoV-2 también genera otros beneficios intangibles, como la educación y la concienciación de la sociedad, o la mejora de la percepción de la población respecto a las acciones institucionales dirigidas a la lucha contra la pandemia. Estos beneficios podrían llegar a justificar su uso, aun a pesar de ser una estrategia con evidencias limitadas o controvertidas sobre su eficiencia.
Comparación exploratoria sobre los costes sanitarios y beneficios de la respuesta de Reino Unido ante la COVID-19 frente a cuatro países europeos
La principal decisión política tomada en distintos países para afrontar la pandemia por SARS-CoV-2 fue el distanciamiento social. Se trata de una estrategia con una repercusión notable, tanto para la población como para la economía del país. Sin embargo, cuantificar el impacto de esta medida puede resultar complicado. Entre las aproximaciones propuestas para evaluar la eficiencia del distanciamiento social, una de las más utilizadas consiste en evaluar los costes, la disminución del Producto Interior Bruto (PIB) y los Años de Vida Ajustados por Calidad (AVACs) perdidos por cada caso de COVID-19, hospitalización o muerte.
En base a esta estrategia, Thom y colaboradores llevaron a cabo una comparativa de la gestión de la pandemia entre Reino Unido, España, Alemania, Suecia e Irlanda. Para cada país se evaluaron dos escenarios: en el primero se tomaron los casos de infección por SARS-CoV-2, hospitalizaciones y muertes publicados, desde el inicio de la pandemia hasta el 20 de julio de 2020; estos se compararon con un segundo escenario en el que se modelizó la transmisión del virus, los casos de hospitalización y las muertes ocasionadas si no se hubieran llevado a cabo las medidas de distanciamiento social. En ambos escenarios se cuantificó el coste asociado a cada episodio de hospitalización del paciente, considerando la frecuencia de hospitalización, los casos que requerían ingreso en UCI y la duración de la estancia, calculando un coste de £4.847 por episodio de hospitalización (4.358€ en 2020, según los datos del Banco Central Europeo). Con vistas a estimar el beneficio clínico neto incremental (BCNI) del distanciamiento social, en el análisis se incluyó exclusivamente el impacto de dicha estrategia sobre la COVID-19 (no se consideró el impacto del aislamiento sobre la salud mental). La eficacia se estimó como AVACs ganados; sin embargo, ante la ausencia de datos específicos sobre SARS-CoV-2, se asumió que el impacto de la infección por SARS-CoV-2 sobre la CVRS era equivalente al impacto de la infección por Influenza. Los AVACs fueron monetizados por medio del umbral de disponibilidad a pagar empleado en Reino Unido (£20.000/AVAC). Finalmente, el impacto económico de la pandemia se estimó mediante las variaciones de los PIB de cada nación en un análisis inicial (Análisis 1). Los resultados obtenidos para cada país fueron comparados entre sí.
Los autores realizaron un segundo análisis (Análisis 2) cuyo fin fue evaluar el efecto de las medidas preventivas tomadas de forma voluntaria por la sociedad. De esta forma, se asumió que los resultados de cada país no tuvieron su origen en las medidas de distanciamiento social tomadas por el Estado, por lo que esta medida no tendría impacto sobre el PIB y, por consiguiente, la disminución de este parámetro sería inferior. Ante la ausencia de un modelo de predicción de las variaciones del PIB en un escenario donde no se aplicasen medidas de distanciamiento social, se tomó como referencia el caso de Suecia, reconocido como el país menos intervencionista en la lucha contra la pandemia, aplicando su disminución del PIB a los otros países.
El análisis de las respuestas gubernamentales ante la pandemia mostró que los países que mayores restricciones impusieron a sus poblaciones fueron Irlanda y España, seguidos de Reino Unido, Alemania y Suecia. Entre estos, España fue uno de los países con mayor número de eventos de infección, hospitalizaciones y muertes evitados (aproximadamente 0,4, 0,05 y 0,03 eventos per cápita, respectivamente). Estos resultados solo fueron equiparables en el caso de Alemania y Reino Unido, siendo destacable el hecho de que Irlanda no alcanzase resultados acordes a su grado de restricción. En esta misma línea, Alemania y España también generaron el mayor BCNI (Tabla 1).
Comparando el Análisis 1 frente al Análisis 2, se puso de manifiesto que la disminución del PIB en España hubiera sido un 51% inferior a la sufrida si se hubiera confiado exclusivamente en las medidas tomadas de forma voluntaria por la población (£2.927,40 frente a £1.727,98). En el caso de Alemania o Reino Unido, la comparación de los dos análisis puso de relieve que el impacto hubiera sido un 46,1% y un 71,10% inferior, respectivamente.
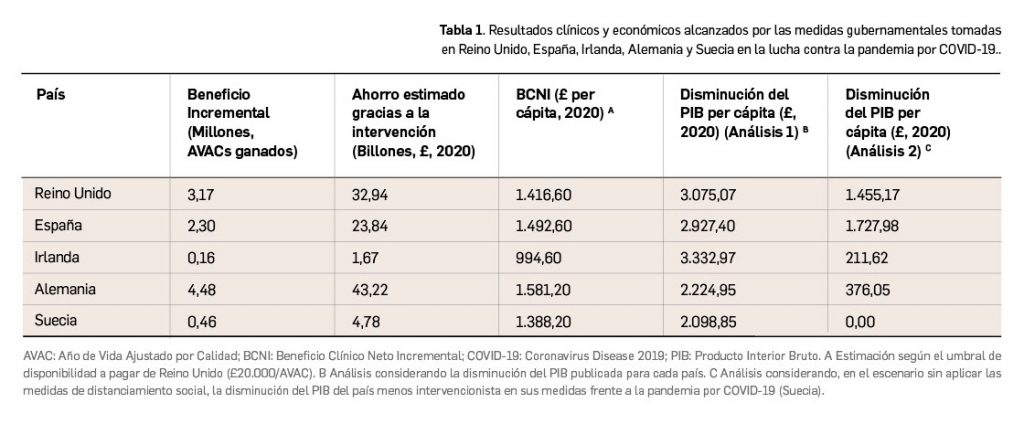
En resumen, Alemania, Reino Unido y España fueron, entre los países comparados, aquellos que alcanzaron mejores resultados (AVACs ganados) en relación con la disminución de sus respectivos PIB. Frente a estos, Irlanda resulta el país con peor balance sanitario y económico tras la pandemia. En el caso concreto de España, las medidas de aislamiento social empleadas evitaron la pérdida de 2,30 millones de AVACs y un ahorro de 24 billones de libras en costes sanitarios, generando un BCNI de £1.493 per cápita. Sin embargo, su PIB se redujo aproximadamente el doble (£3.000 por individuo).
Comentario
Este artículo muestra la gran variabilidad de medidas estatales tomadas para combatir la COVID-19. No obstante, el impacto de la COVID-19 iría más allá del beneficio clínico neto.
Además, el impacto de la pandemia no se ve afectado exclusivamente por la actuación política de cada nación. Los resultados de la comparación entre el Análisis 1 y el Análisis 2 evidencian que las medidas preventivas tomadas de forma voluntaria por la población fueron esenciales en la lucha contra la pandemia, tanto a nivel de infecciones, hospitalizaciones y muertes evitadas, como en términos económicos. De esta forma, concienciar a la población con la prevención de la infección, el uso extendido de la mascarilla o un cribado mayor de pacientes puede resultar más eficaz que apostar por fuertes medidas restrictivas.
Tecnologías ómicas en la investigación de plantas medicinales: nuevas estrategias para la búsqueda de compuestos activos
Resumen
Las plantas medicinales son una importante fuente de metabolitos secundarios, de estructuras muy diversas y con importantes propiedades farmacológicas. Estos metabolitos se originan a través de diversas rutas biosintéticas, las cuales hasta hace poco han sido exploradas de forma limitada. Los recientes avances en los métodos analíticos de alto rendimiento, la capacidad de generación de datos ómicos y el desarrollo de herramientas informáticas para su análisis e integración, han permitido descubrir las vías biosintéticas de los metabolitos activos más relevantes de origen vegetal. Dado que la demanda de compuestos medicinales aumenta con la población mundial, entender la biosíntesis completa de estos compuestos es importante para identificar y/o desarrollar fuentes alternativas y sostenibles de nuevas moléculas con actividad farmacológica. En este artículo se revisan las contribuciones de los principales enfoques ómicos en el estudio de los metabolitos bioactivos y su repercusión en el empleo de plantas medicinales en terapéutica.
Introducción
Las plantas son una fuente de diversos metabolitos especializados que se han utilizado durante miles de años por sus propiedades terapéuticas, pero también como pigmentos, aromatizantes, etc. Estos metabolitos son la herramienta principal utilizada por las plantas para adaptarse y sobrevivir en diferentes nichos ecológicos y condiciones ambientales, así como para contrarrestar las invasiones bióticas en su hábitat. Se estima que en el reino vegetal se producen entre 200.000 y 1.000.000 de metabolitos secundarios diferentes y que más de dos tercios de los fármacos constituidos por moléculas pequeñas introducidos en los últimos 20 años provienen de extractos de plantas o son derivados semisintéticos de los componentes en ellas presentes.
Los metabolitos secundarios de las plantas se caracterizan por poseer una compleja estructura química, que deriva de unidades básicas simples, lo que sugiere la participación en su formación de sofisticados mecanismos biosintéticos y procesos reguladores que han sido favorecidos y mejorados por la selección natural a lo largo del tiempo. Muchos de estos metabolitos presentan bioactividad y toxicidad contra depredadores, proporcionando una ventaja selectiva. Como se ha comentado, las plantas los sintetizan para hacer frente a cualquier amenaza a su crecimiento y supervivencia, pero estos compuestos bioactivos también son útiles para los seres humanos, ya sea por el empleo tradicional para tratar diversas enfermedades de las plantas que los producen, o como base para el descubrimiento y desarrollo de fármacos en la terapéutica moderna.
En este contexto, los enfoques basados en las tecnologías ómicas, fundamentalmente la genómica, la transcriptómica, la proteómica y la metabolómica, junto con las herramientas bioinformáticas disponibles para su análisis e interpretación, han supuesto un importante avance en la caracterización funcional de los genes responsables de la formación de compuestos activos y en la comprensión de los mecanismos de respuesta de los sistemas vegetales. Son fundamentales para el desarrollo de modelos metabólicos y, a su vez, ayudan a diseñar estrategias para generar sistemas bacterianos o vegetales que produzcan compuestos de interés, ampliando la investigación sobre metabolitos secundarios de plantas medicinales y, por tanto, su empleo en terapéutica.
Genómica y transcriptómica en la identificación molecular de especies
Una de las etapas más importantes del descubrimiento de fármacos de origen vegetal es la necesidad de identificar con precisión la especie de la planta de la que procede un compuesto, lo que permitirá atribuir los efectos terapéuticos del mismo a la especie vegetal correcta, evitando así la posibilidad de utilizar una fuente equivocada. Es muy frecuente que las plantas se recolecten directamente de su hábitat natural, por lo que la correcta identificación y nomenclatura son esenciales para su empleo. Para que la identificación sea inequívoca, además de la caracterización morfológica y anatómica tradicionalmente utilizadas, será necesario combinar diferentes métodos, como el análisis genético y el químico, ya que, en muchas circunstancias, los exámenes macroscópicos o microscópicos no serán aplicables, por ejemplo, cuando una preparación consista en muestras de polvo multicomponentes que han sido procesadas perdiendo la posibilidad de la identificación morfológica. Los recientes avances en las técnicas genómicas han permitido establecer un criterio de identificación preciso para las plantas y otras fuentes de productos naturales, como el código de barras del ADN o, en inglés, DNA barcoding, el cual proporciona una identificación rápida y precisa de las plantas en comparación con los métodos morfológicos y otros métodos tradicionales utilizados.
Además, las técnicas genómicas se pueden utilizar para desarrollar diferentes marcadores, incorporándolos en chips genómicos que permiten el uso de métodos de alto rendimiento en la autenticación de fuentes de productos naturales.
DNA Barcoding
Los avances en la secuenciación de ADN han impulsado el empleo del código de barras de ADN (DNA barcoding y metabarcoding) en la identificación y autentificación de plantas medicinales, que posteriormente serán utilizadas en el descubrimiento de fármacos innovadores. Se emplea para verificar una corta secuencia de ADN comparándola con una biblioteca de referencia. Si bien es cierto que la idea de utilizar secuencias de ADN específicas para la identificación rápida de especímenes no es nueva, lo que es innovador del empleo del Código de Barras de ADN es que propone usar información dentro de una misma región génica, en todos los taxones y con condiciones de secuenciación universalmente aceptadas y estandarizada, y a una relación coste-eficiencia relativamente baja. Además, el protocolo destaca la necesidad de relacionar esta información con ejemplares depositados en herbarios.
Estos métodos genómicos se basan en la diversidad de secuencias de regiones cortas y estándar de ADN (400-800 pares de bases) que permiten la identificación precisa y fiable a nivel de especie. El DNA Barcoding consiste, pues, en la utilización de una pequeña región de un gen (denominada DNA Barcode), elegida por consenso científico, para la identificación molecular de la especie; son secuencias relativamente cortas que se obtienen mediante amplificación utilizando la técnica de reacción en cadena de la Polimerasa (PCR). En plantas, se ha propuesto la utilización conjunta de dos regiones de ADN: los genes que codifican para la subunidad mayor de la enzima RuBisCo (Ribulosa-1,5-bisfosfato carboxilasa oxigenasa) (rbcl) y para la maturasa K (matK), localizados en el genoma del cloroplasto (CBOL plant working group, 2009; Consortium for the Barcode of Life), si bien, dependiendo del grupo de plantas que se desee identificar, también se emplean otros marcadores, como ITS, trnH-psbA o ycf5 o una combinación de varios de estos cinco. Combina, por tanto, los métodos basados en la reacción en cadena de la polimerasa (PCR) con las técnicas de secuenciación del ADN.
Para que la técnica sea eficaz y pueda cumplir su objetivo de relacionar una muestra con una especie conocida, es imprescindible contar con bases de datos que incluyan las secuencias de estos marcadores en diferentes especies, lo que permitirá compararlas con las secuencias obtenidas tras la secuenciación y determinar así la especie con la que estamos trabajando. Por esta razón se han creado bibliotecas de libre acceso, siendo las más importantes BOLD (Barcode of Life Database), de la Universidad Guelph en Ontario, creadores de la técnica, y la Base de Datos Colaborativa Internacional de Secuencia de Nucleótidos (International Nucleotide Sequence Database Collaborative), una colaboración entre GenBank (Estados Unidos), Nucleotide Sequence Database (Europa) y DNA Data Bank (Japón). En ambos casos, para el registro de las secuencias obtenidas se deben seguir unas normas acordadas por el CBOL (Consortium for the Barcode of Life).
Desde que se estableció la técnica de DNA Barcoding, sus aplicaciones han ido en aumento e incluyen la identificación tanto de nuevas especies como de especies crípticas, es decir, sin diferencias morfológicas entre ellas y, por lo tanto, sin datos evidentes que permitan distinguirlas.
Actualmente esta técnica es utilizada tanto por la comunidad científica como por la industria para la identificación molecular, con el fin de resolver cuestiones relacionadas con taxonomía, filogenia molecular, genética de poblaciones y biogeografía, así como para evitar la recolección y el comercio ilegal de la fauna y flora silvestres.
Entre las aplicaciones recientes, cabe citar el empleo de la región ITS para identificar adulteraciones de H. perforatum o de la secuencia rbcl para autentificar especies medicinales de la familia Acanthaceae. La técnica también se ha utilizado para la identificación de especies vegetales registradas en diversas Farmacopeas, como la Británica o la Farmacopea Japonesa; por ejemplo, Amaranthus hybridus L., para la que se han utilizado la región ITS2 o la amplificación de la secuencia psbA-trnH.
Estudios realizados aplicando ADN barcoding a 100 muestras de especies vegetales utilizadas con fines medicinales confirmaron que el 59% de los productos incluían muestras de ADN no incluidas en la lista. En la misma línea, estudios realizados para distinguir entre el ginseng coreano y el americano, utilizaron la secuenciación del ADN y la información del código de barras y confirmaron que solo la región ITS es capaz de discriminar adecuadamente entre los distintos tipos de ginsengs. Los autores probaron 95 productos y concluyeron que la mitad del ginseng coreano estaba erróneamente etiquetado como ginseng americano. En cuanto a ADN-Metabarcoding, su mayor ventaja es la capacidad de identificar cada una de las especies dentro de mezclas complejas de múltiples componentes, en las que la aplicación simple del código de barras del ADN y los métodos analíticos convencionales presentan considerables limitaciones.
Una vez que la especie vegetal ha sido autentificada mediante el código de barras de ADN, se pueden desarrollar biotecnológicamente chips de ADN en los cuales se incorporarán los marcadores genómicos, proporcionando una herramienta eficaz para el genotipado. Esta expresión de los genes mediante el análisis de microarray o chips de ADN es una tecnología transcriptómica (presente en una célula o grupo de células en un momento determinado) innovadora que permite un análisis rápido y eficaz de muchas transcripciones y, por lo tanto, de las variaciones en las expresiones de múltiples genes. Se define transcriptoma como el conjunto de todas las moléculas de ARN, también llamadas tránscritos.
Estas tecnologías podrán aplicarse también para descubrir las dianas farmacológicas y dilucidar los mecanismos moleculares de los compuestos y las conexiones biológicas que subyacen a sus acciones farmacológicas y, combinadas con el screening de alto rendimiento, y junto con la disponibilidad de enormes bases de datos de compuestos, lograrán acortar el tiempo necesario en el proceso de descubrimiento y desarrollo del fármaco, desde su diseño hasta los ensayos clínicos.
Son métodos que también tienen limitaciones, pues, aunque pueden proporcionar una autentificación positiva de las especies vegetales basada en la presencia de cualquier ADN amplificable, puede haber falsos negativos si el ADN se ha degradado durante el procesado posterior a la recolección. Además, altas concentraciones de ciertos metabolitos secundarios, como polisacáridos, aceites esenciales, compuestos fenólicos, entre ellos los taninos, o alcaloides pueden interferir en la extracción de ADN o la PCR.
Finalmente, es preciso recordar que, en el contexto del control de calidad de los productos a base de plantas, el ADN barcoding y el metabarcoding no proporcionan ninguna información cuantitativa ni cualitativa de los metabolitos activos de la materia prima vegetal, por lo que su principal aplicación son los procedimientos de identificación y autentificación. Sin embargo, el uso para identificar y discernir taxones en cualquier etapa de desarrollo o procesamiento de los que se puede extraer el ADN es una ventaja esencial para ambos métodos.
Transcriptómica. Secuenciación de ARN
La secuenciación del transcriptoma o secuenciación del ARN (RNA-Seq) es una técnica de alto rendimiento, con alta sensibilidad y resolución, que se emplea para estudiar organismos, por lo que representa uno de los avances más destacados de la biología en los últimos tiempos. Es un método importante para estudiar los genomas de las plantas medicinales, que puede ayudar a analizar los genes funcionales y sus mecanismos de regulación, así como a mejorar las técnicas de selección y cultivo. La investigación y la aplicación de la secuenciación del transcriptoma se centra en el estudio de las siguientes áreas: la extracción de genes funcionales, el desarrollo de marcadores moleculares, las vías biosintéticas que originan los metabolitos secundarios y los mecanismos de desarrollo de las plantas medicinales.
En los últimos años, con los constantes avances en la tecnología de secuenciación, las herramientas de investigación del transcriptoma han evolucionado desde la tradicional plataforma de hibridación en chip a la tecnología de secuenciación de ARN (RNA-Seq), basada en la secuenciación de nueva generación (NGS), que permite la investigación tanto de los genes funcionales como de los genes expresados diferencialmente (DEGs).
Referidas a la investigación de plantas medicinales, la transcriptómica y la genómica tienen varias diferencias clave. En primer lugar, el ensamblaje del genoma es más complejo y costoso que el análisis transcriptómico y, en segundo lugar, hay que tener en cuenta que el transcriptoma se altera en función del tiempo y el espacio de observación, ya que no solo refleja las diferencias en la expresión génica en distintos puntos temporales y espaciales, sino que también incluye información sobre las vías metabólicas que conducen a la formación de los metabolitos secundarios. Diversos estudios han probado que, debido a los diferentes entornos y periodos de crecimiento de las plantas medicinales, incluso entre las mismas especies los patrones de expresión génica reflejan diferencias temporales y espaciales, lo que da lugar a patrones únicos de acumulación de principios activos: así, el transcriptoma es idóneo para identificar los genes relacionados con estos componentes activos.
Mediante el uso de técnicas novedosas, como el análisis de microarrays, es posible analizar de forma eficiente y rápida los tránscritos (secuencias de ARN). A diferencia del análisis de microarrays, el perfil del transcriptoma basado en RNA seq no requiere información genómica previa. Esto permite evaluar los mecanismos de acción de los productos naturales o compuestos aislados, así como los mecanismos moleculares a través de los cuales actúan.
Metabolómica y metabonómica en la identificación de compuestos activos
La metabonómica tiene como finalidad medir la respuesta metabólica global y dinámica de los sistemas vivos a los estímulos biológicos o a la manipulación genética; tiene por objetivo el de comprender el cambio en los sistemas multicelulares complejos a través del tiempo. La metabolómica, por su parte, busca una descripción analítica de muestras biológicas complejas y pretende caracterizar y cuantificar todas las moléculas de la muestra. Estudia, por tanto, el conjunto completo (y dinámico) de los metabolitos que se encuentran en un momento determinado en una célula, tejido u órgano. En la práctica, los términos suelen utilizarse indistintamente.
En el reino vegetal, la diversidad de metabolitos (entendido como compuesto químico o sustancia producida por el organismo) es asombrosa: baste decir que en la base de datos Dictionary of Natural Products (base de datos referencial sobre datos químicos, físicos y estructurales de Productos Naturales) se recogen hasta la fecha aproximadamente 300.000 compuestos de los cuales 200.000 son metabolitos secundarios de plantas, agrupados en 170.000 estructuras, si bien se piensa que el número total de metabolitos producidos por las plantas alcanza el millón, como se ha sugerido anteriormente.
Como hemos avanzado, la metabolómica tiene como objetivo analizar cualitativa y cuantitativamente todos los metabolitos contenidos en un organismo en un momento determinado y en unas condiciones determinadas, elaborando así su denominado perfil metabólico. Sus estrategias actuales dependen principalmente de cuatro enfoques principales: cromatografía de gases (GC-MS), cromatografía líquida (LC-MS), electroforesis capilar (CE-MS), todas ellas acopladas a espectrometría de masas, además de espectroscopia de resonancia magnética nuclear (NMR), con los recientes avances en sus aplicaciones.
La aplicación de la metabolómica en la investigación de los productos naturales comenzó casi una década después de haber sido aplicada en biomedicina y agricultura, y para ello fue decisivo la aparición de los detectores de diodo array y de HRFTMS (espectrometría de masas por transformada de Fourier de alta resolución), los cuales se acoplaron a la cromatografía líquida de alto rendimiento (HPLC).
Para la separación de los metabolitos se suele emplear Cromatografía de Gases o la Cromatografía líquida de alta eficacia (HPLC), y desde principios de este siglo, los avances en los enfoques basados en la Espectrometría de Masas (EM) o la Resonancia Magnética Nuclear (RMN) asociados a las herramientas bioinformáticas han sido decisivos para el diseño y el desarrollo de los estudios de productos naturales. Estos métodos se utilizan para investigar el contenido molecular de los sistemas biológicos con una sensibilidad y precisión sin precedentes. De tal forma que la mayoría de los estudios actuales de perfiles de metabolitos se realizan con herramientas como LC-MS de alta resolución de última generación que combinan la alta resolución de la cromatografía líquida de ultra alto rendimiento (UHPLC) con la espectrometría de masas de alta resolución para la asignación molecular. Si bien la cromatografía suele realizarse mediante la cromatografía de líquidos en fase inversa (RP), otros métodos alternativos se basan en el uso de la cromatografía de líquidos de interacción hidrofílica (HILIC) o la cromatografía de fluidos supercríticos (SFC) para la determinación de los productos naturales muy apolares o lipofílicos.
Para obtener la información precisa final sobre las estructuras que permita la identificación de los compuestos, se emplea la espectrometría de masas acoplada con bases de datos y herramientas informáticas como son Golm metabolome database y Metlin, una base de datos con 62.000 espectros que recoge más de 12.000 metabolitos.
El empleo de estas técnicas ha permitido la identificación de los compuestos responsables de la actividad farmacológica de especies como Newbouldia laevis (P.Beauv.) Seem. y Cassia abbreviata Oliv., antimaláricas, Hyptis suaveolens (L.) Poit., antiinflamatoria y antioxidante, y Panax ginseg C.A. Meyer; concretamente en este último caso, la identificación de una nueva saponina con propiedades antitumorales.
La espectrometría de masas de alta resolución (HRFTMS) y la espectroscopia de RMN se están utilizando también para desreplicar y cuantificar metabolitos conocidos frente a los nuevos productos naturales. La desreplicación combina métodos analíticos que incluyen la espectroscopía ultravioleta, la espectrometría de masas y los datos espectrales de RMN. Los datos de ultravioleta solo pueden utilizarse para el análisis y la desreplicación de los metabolitos que contienen cromóforos, mientras que con la espectrometría de masas existe el riesgo de que haya compuestos pobremente ionizados que solo sean detectables en una forma de ionización. Por ejemplo, los compuestos fenólicos y antraquinónicos se ionizan mal en la modalidad de iones positivos, pero se ionizan muy bien en la modalidad de iones negativos, mientras que lo contrario puede decirse de los alcaloides.
Tales técnicas se han utilizado también en el control de calidad en muestra comerciales como, por ejemplo, para determinar el porcentaje de los diversos componentes de la manzanilla (Matricaria recutita L.), determinar el ácido ∆9-tetrahidrocannabinólico (THC) y el ácido cannabidiólico (CBD) para discriminar entre las diferentes variedades de Cannabis sativa, y determinar los flavonoides con propiedades antidepresivas de distintos lotes del mismo proveedor de Hypericum perforatum. Adicionalmente, se ha aplicado la metabolómica para detectar adulteraciones de preparados de especies similares, por ejemplo, medicamentos antimaláricos que contenían Artemisia afra –que carece de artemisinina– en lugar de Artemisia annua, lo que puede ser fácilmente detectado por 1H-NMR. La metabolómica puede usarse incluso para analizar fluidos biológicos de individuos que han sido tratados con productos naturales más o menos complejos.
Las herramientas de fraccionamiento guiadas por la metabolómica permiten determinar con precisión los componentes activos durante el fraccionamiento de los extractos, así como predecir qué estructuras podrían ser bioactivas, lo que permitiría identificar las fracciones que se someten a purificación, con el consiguiente ahorro de tiempo y recursos durante el proceso.
También los estudios metabolómicos pueden utilizarse para proponer precursores biosintéticos que aumenten el rendimiento del principio activo. Por ejemplo, para la producción del broncodilatador efedrina, la elaboración de perfiles metabólicos específicos y los análisis bioquímicos comparativos revelaron que el benzaldehído es un importante precursor de los alcaloides fenilpropilaminícos producidos por Ephedra spp. U otro ejemplo: mediante la elaboración de perfiles metabólicos fue posible investigar la biosíntesis de tanshinonas y aumentar la expresión de una de las enzimas que intervienen en su síntesis en Salvia miltiorrhiza, concretamente la SmCPS (copalil difosfato sintasa), aumentando la producción de estos principios activos; una de las tanshinonas activas, la IIA tanshinona, ya se utiliza en terapéutica para tratar la isquemia cerebral, presentando también propiedades antiinflamatorias y antitumorales (Majolo et al., 2019).
A principios de este milenio, surgió la secuenciación genómica de alto rendimiento, lo que permitió pasar de la investigación genética pura al estudio de la función y expresión de los genes. No podemos olvidar que la capacidad de un organismo para producir metabolitos secundarios es un carácter fenotípico. Este enfoque permite el estudio indirecto de la función de los genes y la bioquímica de un organismo. Además, se puede utilizar una combinación de metabolómica y genómica para optimizar una ruta biosintética a fin producir selectivamente metabolitos secundarios biológicamente activos.
Proteómica en la validación e identificación de biomarcadores
El análisis proteómico ha surgido como un enfoque complementario a los ya mencionados genómicos y transcriptómicos, aplicado a la identificación de los mecanismos de acción de muchos productos naturales. A partir de la expresión de proteínas, su función y biosíntesis, la proteómica también puede reflejar la calidad del producto natural. Los cambios asociados con el crecimiento de la planta o en respuesta a factores ambientales se estudian utilizando técnicas proteómicas (Ghatak et al., 2017; Hashiguchi et al., 2017), como también las proteínas específicamente relevantes para el control de las vías metabólicas, permitiendo, por ejemplo, acelerar el desarrollo de los cultivos.
Así pues, la investigación proteómica tiene como objetivo comprender la expresión y las funciones de las proteínas a nivel global y requiere integrarse con otra información, como los perfiles de los genes, los ARNm y los metabolitos, para comprender plenamente el funcionamiento de los sistemas biológicos. Las tecnologías proteómicas se han utilizado para estudiar los efectos farmacológicos y los mecanismos de acción de las plantas medicinales, para identificar moléculas diana o incluso para identificar nuevos componentes bioactivos.
Un ejemplo ilustrativo es la aplicación de análisis proteómicos en investigaciones sobre Panax ginseng, utilizada en medicinas tradicionales para tratar trastornos como la diabetes, las enfermedades cardiacas, el cáncer y la neurodegeneración. Sus principios activos, los ginsenósidos, son saponinas triterpénicas que se encuentran casi exclusivamente en Panax spp. (Leung et al., 2010). Dado que el cultivo del ginseng es difícil por su largo ciclo de vida, resulta crucial comprender las vías biosintéticas por las que se originan estos compuestos (Liang et al., 2008). Los análisis proteómicos aplicados a estudios de crecimiento y desarrollo de la planta revelaron que las raíces de ginseng inician la biosíntesis de ginsenósidos cuando la planta alcanza un periodo de crecimiento lento. Esos cambios en la fisiología de las raíces demuestran la actividad temprana de las enzimas que intervienen en los procesos biosintéticos de ginsenósidos. En línea con esto, el reciente análisis proteómico de un tipo de ginseng cultivado que crece más lentamente que el ginseng silvestre, reveló que éste aumenta la biosíntesis de ginsenósidos a medida que la planta envejece. El estudio del perfil metabólico permitió establecer la correlación de los cambios energéticos en el metabolismo con el contenido de ginsenósidos (Liu et al., 2017).
La proteómica también se emplea en estudios relacionados con la actividad. Por ejemplo, también en el ginseng, estudios proteómicos han permitido establecer que existe una correlación entre la cantidad de una proteína de la familia de la 6-fosfogluconato deshidrogenasa y la actividad captadora de radicales libres de muestras de ginseng provenientes de diferentes cultivos (Kim et al., 2016).
Con respecto a las técnicas empleadas para el análisis proteómico del metabolismo secundario de las plantas medicinales, destacan entre las que han resultado más eficaces la purificación de las proteínas poco abundantes mediante la aplicación de bibliotecas combinatorias de ligandos peptídicos (CPLL) o el tratamiento de fraccionamiento de polietilenglicol (Zhang et al., 2013; Zhang et al., 2014).
Es amplia la evidencia que muestran la utilidad de las técnicas proteómicas para descubrir enzimas que son cruciales para actividades biológicas de las plantas medicinales, siendo también eficaces para estudiar las redes del metabolismo secundario y las nuevas enzimas que permanecen inexploradas. Así, ha sido aplicada al estudio del contenido en metabolitos secundarios de Catharanthus roseus, concluyendo que el mismo mejoró gracias a un incremento de la enzima 10-hidroxigeraniol oxidorreductasa, de la 1-deoxi-d-xilulosa 5-fosfato reductoisomerasa y la 5-enol-piruvilshikimato-fosfato sintasa en Lonicera japonica.
Conclusiones
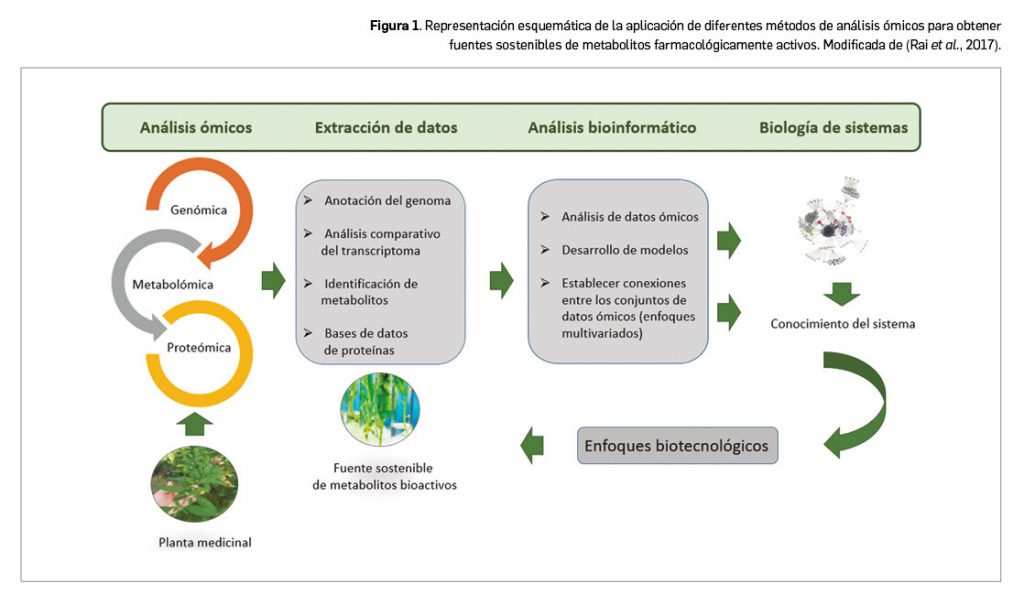
Las tecnologías ómicas se han desarrollado en respuesta a la necesidad de disponer de estrategias de biología molecular para el estudio de organismos vivos (Figura 1). Aplicadas al estudio de productos naturales aportan información amplia y coherente que permite obtener compuestos bioactivos. Actualmente se aplican en diferentes etapas de la investigación de plantas medicinales, desde la estandarización, el control de calidad de las formulaciones vegetales y la identificación de los mecanismos moleculares hasta la predicción de los efectos secundarios y las interacciones con otros medicamentos. Los datos obtenidos con el empleo de estas técnicas permiten descubrir vías metabólicas y enzimas no identificadas, genes, redes de genes, nuevos metabolitos e interacciones proteína-proteína.
Los estudios sobre la biología de sistemas aplicando la genómica, la transcriptómica, la metabolómica y la proteómica a la investigación interdisciplinar de productos naturales constituyen, por tanto, una valiosa herramienta para encontrar nuevos metabolitos secundarios biológicamente activos. Albergan también potencial para mostrar las complejas respuestas farmacológicas de los preparados vegetales. Por todo ello, contribuyen a que la gran diversidad biológica de las plantas se pueda utilizar para el descubrimiento de nuevos fármacos.
Insuficiencia cardiaca y su papel en el eje cardio-renal-metabólico
Según la Sociedad Europea de Cardiología (ESC), la insuficiencia cardiaca (IC) se define como un síndrome clínico caracterizado por síntomas típicos (dificultad para respirar, hinchazón de los tobillos y fatiga), que puede acompañarse de signos (elevación de la presión venosa yugular, estertores pulmonares y edema periférico), causada por una anomalía cardiaca estructural y/o funcional, que resulta en un gasto cardiaco reducido y/o presiones intracardiacas elevadas en reposo o durante el estrés.
Los signos y síntomas de la IC se pueden agrupar de la siguiente manera:
- Reducción del gasto cardiaco (astenia, cansancio, disnea).
- Aumento de la presión capilar pulmonar (falta de respiración).
- Sobrecarga de volumen extracelular (edemas).
Se trata de una enfermedad relevante en la salud mundial cuya prevalencia es del 1-2% en países desarrollados: se estima que 26 millones de pacientes viven con IC. En España, la prevalencia alcanza el 5% y supone, por tanto, un gran impacto sociosanitario. A diferencia del resto de patologías cardiacas, cuya evolución, prevalencia e incidencia ha ido mejorando a lo largo de los años, esta enfermedad va aumentando de forma progresiva y la tendencia es que siga haciéndolo. Entre las razones se encuentran el progresivo envejecimiento de la población (a mayor envejecimiento de la población, mayor probabilidad de desarrollar IC), el aumento de la supervivencia de pacientes con problemas cardiacos, el aumento de los factores de riesgo cardiovascular (hipertensión, diabetes), así como la mejoría del pronóstico y manejo de la IC.
Esta patología también presenta una alta morbilidad, reduciendo la calidad de vida y el deterioro funcional de los pacientes e incluso de los familiares. Además, es la causa más frecuente de ingresos hospitalarios, la cuarta causa de mortalidad en nuestro medio y supone un 3,8% del gasto sanitario.
Relación entre diabetes e insuficiencia cardiaca
Existe una estrecha relación entre la IC y la diabetes mellitus tipo 2, de manera que los diferentes ensayos clínicos han demostrado que los pacientes con diabetes tienen mayor riesgo de sufrir IC y viceversa.
Son diversos los mecanismos que interfieren en esta conexión cardio-metabólica. Entre ellos se encuentran el estrés oxidativo, la inflamación, la fibrosis, la aterosclerosis, la hipertensión, etc., que llevan a una alteración de la perfusión y a la presencia de enfermedad coronaria, hipertrofia, apoptosis, aumento de la utilización de la glucosa y a la alteración de la función mitocondrial, entre otras. Todo ello provoca una disfunción sistólica o diastólica y, en consecuencia, la aparición de IC diabética (miocardiopatía diabética).
Relación entre enfermedad renal e insuficiencia cardiaca
Por su parte, el corazón y el riñón son órganos muy relacionados entre sí: producen un continuo cardio-vásculo-renal que explica que la afectación cardiaca dé lugar a la afectación renal o viceversa (síndrome cardio-renal).
La insuficiencia renal (IR) es muy frecuente en pacientes con IC y empeora el pronóstico de esta. Tanto la albuminuria como la reducción de la tasa de filtración glomerular son marcadores pronósticos de la IR. Además, la albuminuria constituye a su vez un factor pronóstico para la IC. A medida que aumenta el deterioro de la función renal y la excreción urinaria de albúmina, se produce un aumento progresivo de las enfermedades cardiovasculares (CV) y, por consiguiente, del riesgo de mortalidad, siendo esta superior en los pacientes diabéticos que en los no diabéticos.
Abordaje farmacoterapéutico y recomendaciones de las guías
El algoritmo terapéutico depende del tipo de IC que presente el paciente. Cuando la fracción de eyección es reducida (< 40 %), el algoritmo propuesto por las guías ESC hasta el año 2016 establecía el tratamiento con un inhibidor de la enzima convertidora de angiotensina (IECA; como enalapril) o con un antagonista del receptor de angiotensina II (ARA-II; como candesartán) y un betabloqueante (por ejemplo, metoprolol, bisoprolol, carvedilol o nebivolol) como tratamiento de primera línea. Si el paciente seguía sintomático, se debía añadir un antagonista del receptor mineralocorticoide (por ejemplo, eplerenona o espironolactona). Si tras este tratamiento el paciente continuaba con síntomas, se proponía cambiar el IECA o el ARA-II por un inhibidor del receptor de angiotensina/neprilisina (ARNI; sacubitrilo/valsartán), mejorando así el pronóstico. Por último, si el paciente estaba en ritmo sinusal y el QRS era > 130 ms, se planteaba la opción de resincronización y, si este entraba en ritmo sinusal y la frecuencia cardiaca era > 70 lpm, la opción era ivabradina.
Sin embargo, la introducción en 2017 de los inhibidores del cotransportador sodio/glucosa de tipo 2 (iSGLT2; empagliflozina, dapagliflozina, canagliflozina y ertugliflozina) ha supuesto un cambio en las guías terapéuticas. Se trata de fármacos antidiabéticos que han demostrado una mejoría notable del pronóstico de los pacientes con enfermedad CV previa, sobre todo en prevención secundaria. Los estudios DAPA-HF (2019, dapagliflozina) y el EMPEROR-Reduced (2020, empagliflozina) han probado que estos fármacos también son útiles para mejorar el pronóstico de IC independientemente de la presencia o no de diabetes, razón por la cual se incorporaron al arsenal terapéutico de la IC. Empagliflozina demostró mejorar también la función renal, demostrando así aún más la relación cardio-renal. Con todos estos resultados, las guías ESC de 2021 establecieron como elección preferente el tratamiento combinado de inicio con IECA/ARNI + betabloqueante + antagonista del receptor mineralocorticoide + iSGLT2.
Por otro lado, las guías ESC 2021 recomendaban, para los pacientes con fracción de eyección preservada, solo los diuréticos como tratamiento para los síntomas. Sin embargo, a la vez que se publicaban estas guías, se llevó a cabo un estudio (EMPEROR-PRESERVED) con un iSGLT2 (empagliflozina), que demostró que este fármaco mejoraba también el pronóstico de los pacientes con fracción de eyección preservada.
En resumen, con el fin de conseguir que el paciente con IC esté óptimamente tratado, se necesita un programa de atención multidisciplinar para reducir su riesgo de hospitalización y mortalidad; dicho programa debe contar, de forma óptima, con la colaboración de todos los profesionales sanitarios (médicos, enfermeros, farmacéuticos, etc.), para poder conseguir un mejor abordaje de la IC.
Nuevo BOT PLUS. Diseño y funcionalidades
Resumen
BOT PLUS, la base de datos de medicamentos y productos de parafarmacia del Consejo General de Colegios de Farmacéuticos, es una herramienta en continua evolución, con el objetivo de cubrir las necesidades informativas del farmacéutico, de forma que le sirva como una herramienta de ayuda en su ejercicio profesional diario. Por este motivo, el Consejo General lanza en 2022 una nueva versión de BOT PLUS, en la que se ha rediseñado completamente la aplicación, con una modernización de la imagen que va a contribuir a que sea más compatible con los dispositivos electrónicos actuales y a mejorar su manejabilidad, simplificando la misma.
______________________________________________________________________________
BOT PLUS, la base de datos de información sanitaria del Consejo General de Colegios de Farmacéuticos, es una herramienta en continua evolución que busca adaptarse a las necesidades cada vez más exigentes de los farmacéuticos en cuanto a información de medicamentos y productos de parafarmacia.
Durante los últimos años se ha trabajado especialmente en la infraestructura tecnológica que sustenta esta herramienta, avanzando igualmente en la profundidad de sus contenidos. La evolución de 2022 se centra en el rediseño completo de la aplicación, con una nueva imagen de BOT PLUS, siguiendo la misma línea de la nueva imagen del Consejo General, y haciéndola más compatible con los dispositivos electrónicos actuales, especialmente smartphones.
Este nuevo diseño va acompañado de una mejora en la manejabilidad de la aplicación, implementando cambios en la funcionalidad de BOT PLUS, con el objetivo de simplificar y modernizar el manejo de los buscadores de la herramienta, haciéndola más intuitiva y sencilla de manejar, y por tanto, aumentando su utilización por parte del usuario.
En este artículo vamos a repasar estas dos principales novedades que se incluyen en esta nueva versión rediseñada de BOT PLUS, y que serán revisadas en mayor profundidad en siguientes números de la revista.
Rediseño de la aplicación
BOT PLUS ha sufrido una renovación en el diseño gráfico, asemejándose a la nueva imagen del Consejo General, modernizando su aspecto externo. Asimismo, se ha trabajado en mejorar la visualización de BOT PLUS a través de dispositivos como teléfonos inteligentes (smartphones) o tablets, cambiando el diseño de la aplicación para adaptarla a un diseño responsive, perfectamente adaptado al tamaño de pantalla, de forma que el uso de BOT PLUS en estos dispositivos móviles se optimice.
Mejoras en la manejabilidad de BOT PLUS
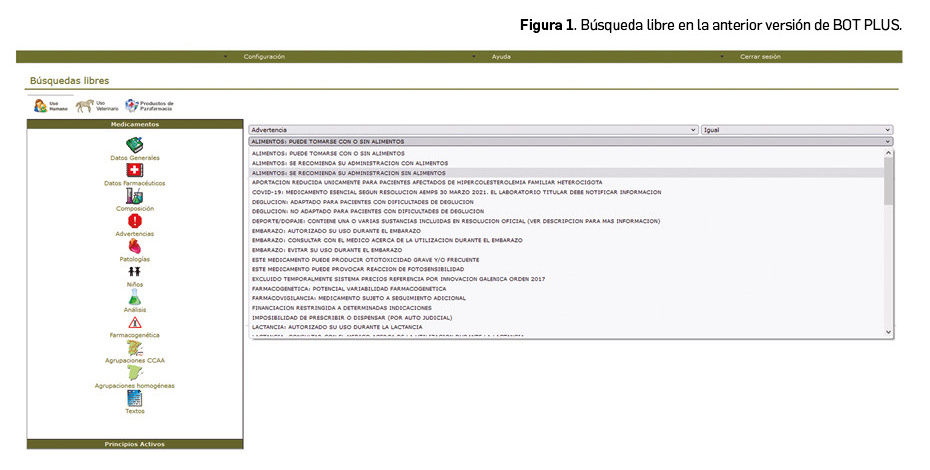
Hasta el momento, BOT PLUS recogía mucha información y, en ocasiones, el manejo de esta información en algunos buscadores era muy complejo, especialmente en la “Búsqueda libre”. Por ello, la principal novedad incluida este año es el rediseño de la aplicación que ha conseguido facilitar el funcionamiento de esta, haciéndola más accesible y, por lo tanto, más útil para los usuarios.
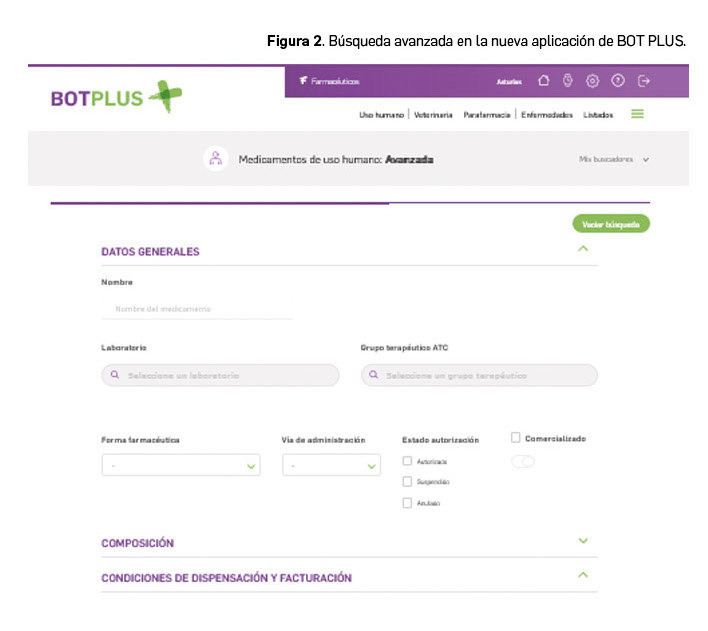
En este nuevo BOT PLUS hemos pasado de los combos desplegables, que caracterizaban la anterior Búsqueda libre (Figura 1) y para cuyo uso se requería estar muy familiarizado con la herramienta (saber en qué carpeta se incluía cada dato), a los nuevos desplegables que incluyen las diferentes informaciones de interés y que en la mayoría de los casos se pueden seleccionar con solo un clic, tal y como se observa en la nueva búsqueda Avanzada (Figura 2).
Ampliación de las funcionalidades existentes en la pantalla de inicio
Se ha renovado totalmente la pantalla de inicio de BOT PLUS, haciéndola más accesible y dinámica, y permitiendo la búsqueda de forma sencilla y rápida de medicamentos y productos de parafarmacia. En esta nueva pantalla de inicio (Figura 3) sigue disponiéndose de un buscador central, con la novedad de ser predictivo, a través del cual se permite seguir buscando medicamentos, principios activos, productos de parafarmacia o enfermedades –por código nacional o nombre–, y se pueden definir los buscadores preferidos. Entre las nuevas funciones que incluye esta nueva pantalla de inicio también destacan:
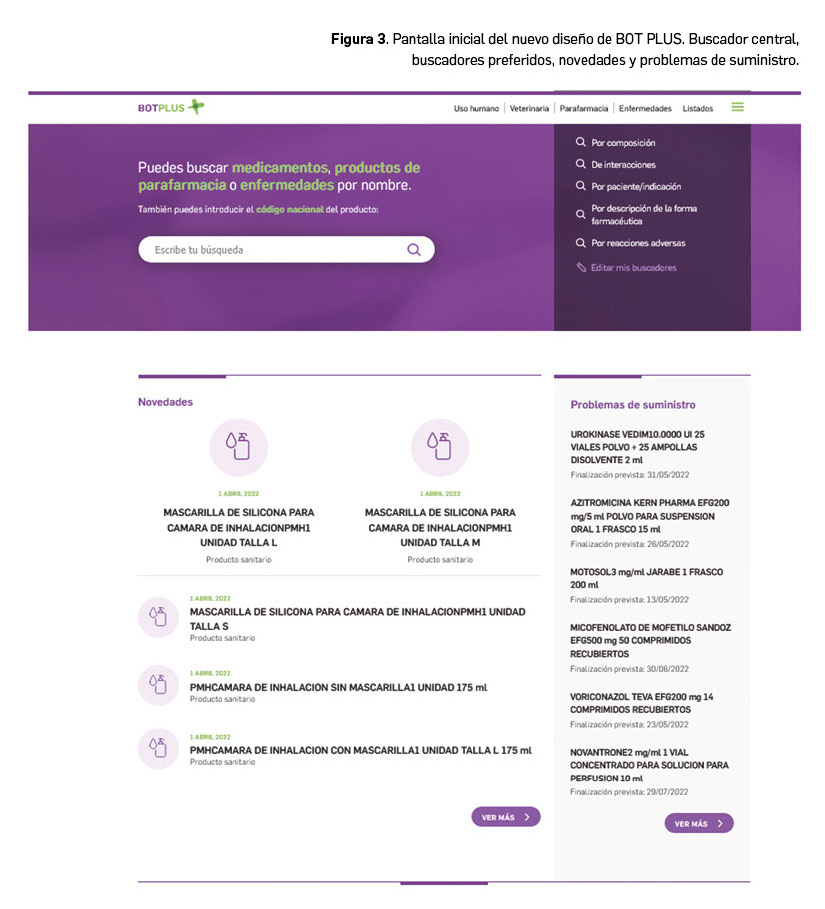
- Últimos productos incluidos. Figuran los últimos registros (medicamentos de uso humano, veterinario o productos de parafarmacia) que se hayan dado de alta en BOT PLUS, de más actuales a más antiguos; si se desean ver más resultados se puede pinchar en el botón “Ver más”.
- Problemas de suministro. Aparecen los últimos medicamentos con problemas de suministro activos publicados por la AEMPS, y al pinchar sobre “Ver más” accederemos a todos los problemas de suministro activos ordenados del más reciente al más antiguo.
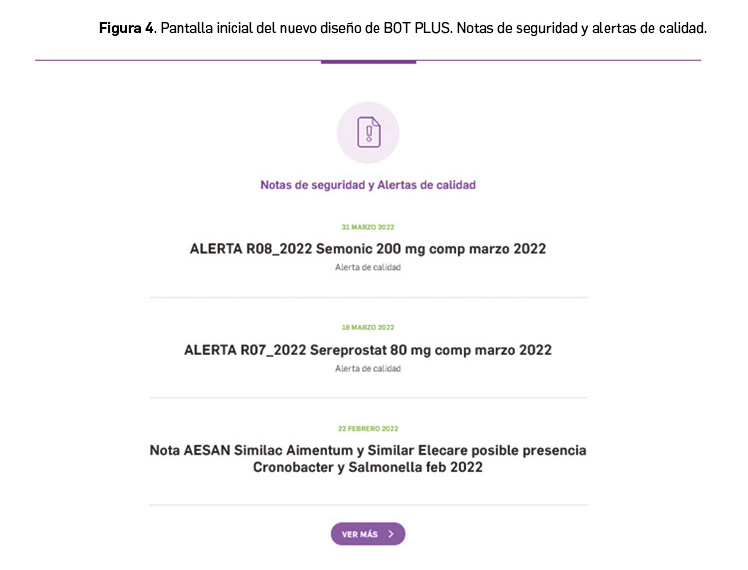
- Notas de seguridad y alertas de calidad. Figuran los últimos documentos de interés profesional para el farmacéutico (Figura 4) de entre los siguientes tipos: alerta calidad AEMPS; comunicaciones riesgo AEMPS veterinaria; notas informativas AEMPS de medicamentos y productos de parafarmacia; notas informativas AESAN; notas seguridad AEMPS.
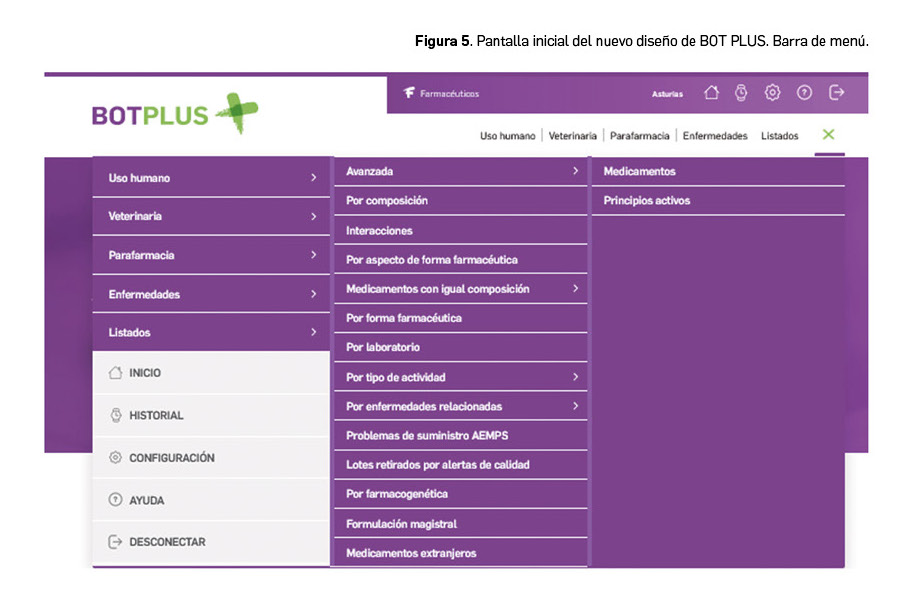
- Inclusión de una barra de menú. Se ha potenciado el menú para hacerlo más visible. Los diferentes accesos que habrá en el menú son: medicamentos uso humano (Figura 5); medicamentos veterinarios; productos de parafarmacia; listados; configurar provincia; ayuda; historial; cerrar sesión.
- Capacidad de personalización
El usuario puede seleccionar como favoritos aquellos buscadores que más utilice, predefiniéndolos y teniéndolos fácilmente accesibles desde la pantalla de inicio. - Potenciación de las funcionalidades de generación de listados
Los listados obtenidos a través de BOT PLUS son dinámicos a partir de ahora, pudiendo al usuario establecer diferentes filtros sobre el listado obtenido, de forma que pueda restringir sobre la marcha las condiciones de búsqueda establecidas, como se verá en los próximos números de Panorama. - Modificación de las fichas de medicamentos y productos de parafarmacia
Las fichas de los medicamentos y los productos de parafarmacia se han modificado para destacar de forma significativa las condiciones de dispensación, financiación y precios de los mismos, de forma que sirvan como una ayuda práctica durante la dispensación. - Ampliación de los listados predefinidos
En esta nueva versión de BOT PLUS se han incluido nuevos listados predefinidos de la herramienta, para obtener información de interés a través de un único clic. A estos listados se puede acceder desde el menú de la barra literal y destacan los siguientes:
- Productos con cambio de nombre.
- Productos con cambios de laboratorio.
- Productos con cambios de precios.
- Medicamentos desfinanciados.
- Medicamentos con precio notificado.
- Medicamentos afectados por las deducciones del RDL 8/2010.
- Medicamentos sujetos a seguimiento adicional.
- Medicamentos obligados a llevar dispositivo de seguridad.
- Medicamentos de anotación en libro recetario o libro estupefaciente.
Este rediseño se ha incorporado en la nueva versión del programa, publicada en este mes de junio de 2022. En definitiva, en las últimas versiones de BOT PLUS siempre se ha mejorado y ampliado la información de medicamentos y productos de parafarmacia con nuevos contenidos, pero en este caso, se ha optado por rediseñar la aplicación para mejorar su visualización a través de dispositivos como smartphones o tablets y facilitar su manejabilidad y funcionamiento, haciéndola más accesible y, por lo tanto, más útil para los usuarios, a fin de responder a las necesidades del farmacéutico para facilitarle un ejercicio asistencial completo.


